DNAシーケンスの重要性
DNADNAは 「deoxyribonucleic acid」の略で(デオキシリボ核酸)、2本のポリヌクレオチド鎖からなるポリマーで、互いに巻きついて二重らせんを形成しています。 More は、すべての生物の遺伝暗号を含む分子であり、DNA内の特定のヌクレオチド塩基配列が生物の生物情報を決定しています。DNAの塩基配列を決定し、DNA分子内のヌクレオチドの順序を決定することで、ヒト、植物、動物、微生物コミュニティの遺伝子機能と全体的なゲノムを理解することができます(Novogene、2011)。DNA配列決定の広告は、生物学および医学研究に大きな影響を与えます(Rothberg et al.、2011)。
第一世代のDNAシーケンス
1970年代後半にケンブリッジ大学のFrederick Sangerらによって開発されたのが、第一世代シーケンス( サンガー配列決定インビトロ DNA 複製中の DNA ポリメラーゼによる鎖終結ジデオキシヌクレオチド (ddNTP) の選択的取り込みに基づく DNA 配列決定技術。 More 、Sanger Sequencing)です。サンガー配列決定は、増幅と ポリアクリルアミドゲル電気泳動ポリアクリルアミドゲルの細孔に電流を流し、分析物を分離する方法。 More を組み合わせたゲルベース法です。このプロセスは、鋳型DNA、DNA プライマーDNA合成を開始するための短い一本鎖核酸。 More 、非標識 ヌクレオチド窒素塩基と糖およびリン酸基が結合した化合物。ヌクレオチドはDNAなどの核酸の基本構造単位を形成します。 More ( デオキシヌクレオチド 三リン酸、deoxynucleotide triphosphates、dNTPs)、塩基ごとに異なる色で蛍光標識した ジデオキシヌクレオチドサンガー配列決定で使用される DNA ポリメラーゼの鎖伸長阻害剤。 More (dideoxynucleotides、ddNTPs)をポリメラーゼ駆動の反応中に混合することで行われます(図1a)。この反応において、ddNTPはDNA鎖の伸長に必要な3’OH基を持たないため、鎖のターミネーターヌクレオチドとして作用し、それ以上の伸長を阻止します。そのため、ddNTPがランダムに組み込まれ、鎖を終結させた異なる長さの鎖が生成されます(図1b、c)。その後、電気泳動により、伸長産物は1塩基分の大きさの違いに分解されて分離されます。鎖が終結した断片は、蛍光ラベルで検出され、それぞれの色が終結したddNTPの1つを識別します(図1d、e)。テンプレートDNA鎖の配列を解析し、再構築することができます(図1e)(Heather & Chain、2016)。このプロセスにより、500~1,000 bpの断片の配列決定が可能になります(Novogene、2011)。

図1. サンガーシーケンスの模式図(BioRender, 2020から改変)。
第2世代シーケンス (Next generation sequencing、NGS)
第一世代シーケンスは年々改良されてきましたが、単位時間あたり少量のDNAしか配列決定できない(=低スループット)ため進歩が少なく、コストが高い(=塩基配列決定あたりのコスト)という欠点を抱えています(Choudhuri, 2014)。そこで、スループットを向上させ、シーケンスにかかるコストを下げるために、第2世代または次世代シーケンス次世代シーケンス(NGS、Next generation sequencing) は、ゲノム全体、あるいはDNAやRNAの標的領域のヌクレオチドの順序を同時に決定できる強力な技術です。 More技術が導入されました。
第2世代のDNAシーケンスは、ライフサイエンス社のRoche 454プラットフォーム、イルミナ社のプラットフォーム、アプライドバイオシステムズ社のSoLiDプラットフォームに代表されますが、本テクニカルノートではイルミナ社のプラットフォームについてのみ詳細に説明します。イルミナのSBS(Sequencing by synthesis)テクノロジーは、サンガーシーケンスと比較して、1回の実行で同時に多くのDNA断片を超並列的にシーケンスすることができます。SBSテクノロジーは、まずシーケンス ライブラリーNGSを行う場合に創るやつ。 More 、または末端に アダプター他の DNA または RNA 分子の末端に連結できる、化学合成された短い一本鎖または二本鎖のオリゴヌクレオチド。 More を取り付けた200~500 bpの一本鎖DNA断片のプールを準備します(図2a)。DNA断片は、アダプターに相補的なフローセル上の表面固定 オリゴヌクレオチドヌクレオチド数が少なく、短くて直鎖状のDNAまたはRNA More (オリゴ)とハイブリダイズすることができます(図2b)。鎖の最初のPCR増幅(図2c、d)、その後のブリッジ( PCRPCR(Polymerase chain reaction、ポリメラーゼ連鎖反応)は、変性、オリゴヌクレオチドのアニーリング、および DNA ポリメラーゼ伸長のサイクルを繰り返すことによる核酸配列の酵素的増幅を伴う技術です。 More )増幅(図2e-g)により、一本鎖のクラスターが生成されます(図2h)。次に、蛍光性可逆ターミネーター化学を使用して、一本鎖の配列決定が繰り返されます。最初のサイクルでは、化学修飾されたヌクレオチドが自然相補性によってDNA鋳型鎖に結合します。各ヌクレオチドには蛍光タグと、次の塩基の取り込みを阻止する可逆的ターミネーターが含まれています(イルミナ、2023年)。各サイクルにおいて、1つのヌクレオチドが組み込まれた後、レーザー励起を受け、放出された蛍光が検出されます。蛍光シグナルは、どのヌクレオチドが付加されたかを示します。ターミネーターは切断され、次の塩基の結合を可能にします(図2i)。このサイクルを繰り返し、各フラグメントの塩基配列を1塩基ずつ決定します(図2i)。順方向のDNA鎖を読み取った後、読み取った塩基を洗い流し、逆方向のDNA鎖についてこのプロセスを繰り返します。シーケンスが完了すると、すべての配列がアラインされ、解析されます(Choudhuri、2014)。このプロセスにより、50~500bpの断片の配列決定が可能とります(Novogene、2011)。
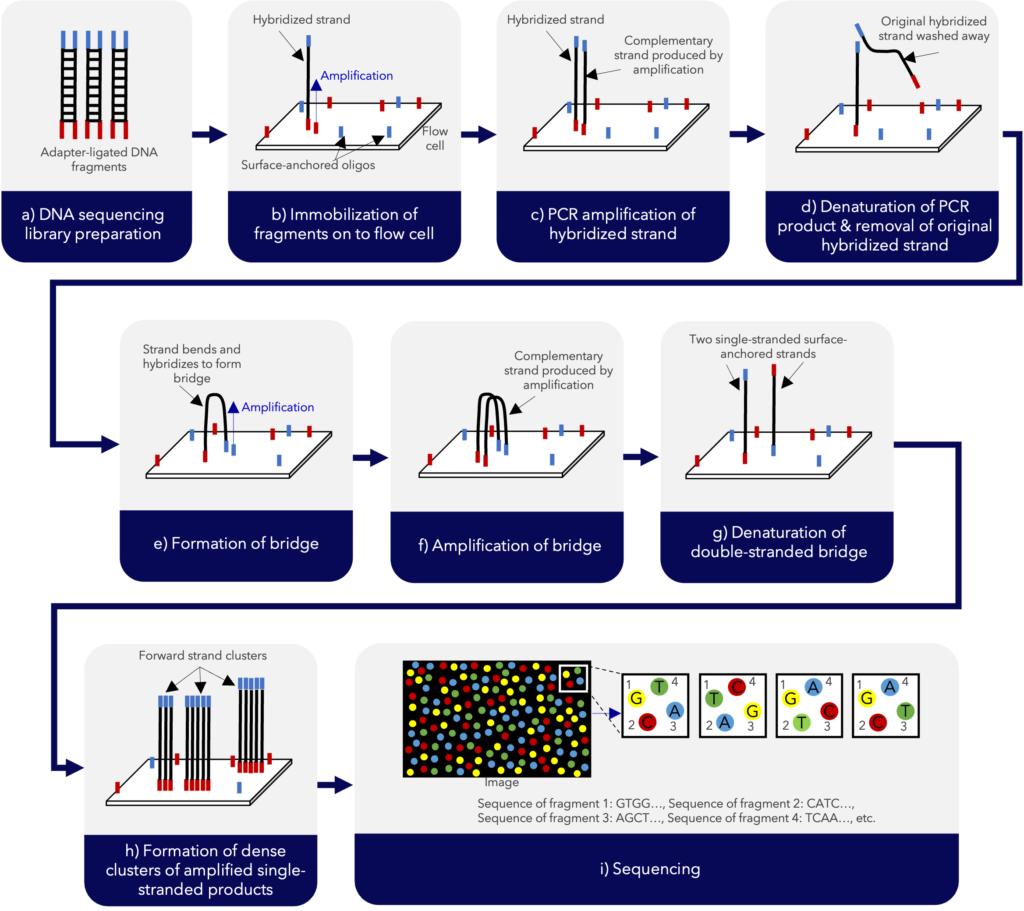
図2. IlluminaのNGSワークフロー(Choudhuri, 2014から改変)。
第3世代DNAシーケンス
第2世代シーケンスは、第1世代シーケンスが抱えていた2つの大きな問題(低スループット、高コスト)を解決した技術であるが、同時に技術的な問題も抱えています。そのような技術的問題の例としては、PCRで生成されたDNAシーケンスライブラリに見られるPCRによるバイアスやエラー、非効率的な蛍光ヌクレオチド標識、蛍光駆動ベースコールの固有のノイズによる1分子蛍光の検出の高いエラーレートなどがあります(Choudhuri、2014)。そこで第3世代シーケンスは、第2世代シーケンスが抱える技術的問題を解決することを目的として開発され、1分子シーケンス技術、PCR増幅なし、複雑なサンプル調製なし、塩基導入後のシーケンス停止なし(シーケンス速度向上)、リード長DNA 断片から配列決定された塩基対 (basepair、bp))の数。 More増加、コスト低減などが盛り込まれています(Choudhuri、2014)。第3世代のDNAシーケンスは、Pacific Biosciences社(PacBio)とOxford Nanopore社のシーケンスプラットフォームに代表されるが(表1)、ここではPacBio社の単一分子リアルタイム(Single-molecule real-time、SMRT)シーケンス技術のみを説明します。
SMRTシーケンス技術は、SBSアプローチを採用し、ポリメラーゼによるDNAの一本鎖の合成をリアルタイムで直接観察することができる技術です。この技術では、数万本のゼロモード導波路(Zero-mode waveguides、ZMW)を含むSMRTセルを使用します。ZMWは、ガラス基板上に蒸着された100nmの金属膜に作られた小さな部屋(直径数十ナノメートル)です。ZMWチャンバーは非常に小さいため、可視レーザー光は完全に通過することができず、レーザー光はZMWチャンバーの底部30nmを照射することしかできません(図3a)。シーケンシング中、DNA断片上のアダプターは、ZMWの底部に固定化されているポリメラーゼに結合し、そこで光の励起と発光が行われます(図3a)。4つのヌクレオチドはそれぞれ異なる蛍光色素で標識されており、ヌクレオチドがポリメラーゼと相互作用することで光パルスが発生します(図3b)。取り込まれた塩基は蛍光発光に基づいて認識され、1つのDNA分子の合成を直接記録することができます(Choudhuri、2014; Huo et al.、2021)。

図3. SMRTシーケンスの模式図(Huo et al、2021から改変)。
DNAシーケンスのアプリケーションについては、こちらをご覧ください。
参考文献
BioRender. (2020). Sanger sequencing. BioRender. https://app.biorender.com/biorender-templates/t-5ef132f6c7bcd500b388a9c3-sanger-sequencing
Choudhuri, S. (2014). Bioinformatics for beginners: Genes, genomes, molecular evolution, databases and analytical tools. Elsevier Inc. https://www.sciencedirect.com/book/9780124104716/bioinformatics-for-beginners
Heather, J. M., Chain, B. (2016). The sequence of sequencers: the history of sequencing DNA, Genomics, 107(1): 1-8. doi: org/10.1016/j.ygeno.2015.11.003. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4727787/
Huo, W., Ling, W., Wang, Z. et al. (2021). Miniaturized DNA sequencers for personal use: unreachable dreams or achievable goals. Front Nanotechnol, 3. doi: org/10.3389/fnano.2021.628861. https://www.frontiersin.org/articles/10.3389/fnano.2021.628861/full
Illumina. (2023). Main steps in next-generation sequencing. Illumina, Inc. https://www.illumina.com/science/technology/next-generation-sequencing/beginners/ngs-workflow.html
Novogene. (2011). A beginner’s guide to DNA sequencing. Novogene Co., Ltd. https://www.novogene.com/us-en/resources/blog/a-beginners-guide-to-dna-sequencing-blog/
Rothberg, J., Hinz, W., Rearick, T., et al. (2011). An integrated semiconductor device enabling non-optical genome sequencing. Nature, 475: 348–352. doi: org/10.1038/nature10242. https://www.nature.com/articles/nature10242
